精準(zhǔn)醫(yī)療,也被稱為個(gè)性化醫(yī)學(xué),是指使用病人特定的分子特征來指導(dǎo)疾病診斷�����、監(jiān)測(cè)病情發(fā)展與治療應(yīng)答、或用于確定藥物開發(fā)新靶點(diǎn)的醫(yī)療模式���。
精準(zhǔn)醫(yī)療原則的應(yīng)用使癌癥治療領(lǐng)域取得了長(zhǎng)足的進(jìn)步���,并有望顯著改變多種病癥的治療,包括代謝和心血管疾病�、自身免疫和炎癥性疾病,以及神經(jīng)發(fā)育和神經(jīng)退行性疾病����。在這篇文章里,我們將回顧精準(zhǔn)醫(yī)療的基本理念和面臨的挑戰(zhàn)���,并討論表觀基因組和染色質(zhì)生物標(biāo)志物如何成為推進(jìn)這一重要研究的關(guān)鍵�����。
?
精準(zhǔn)醫(yī)療的必要性
就歷史觀點(diǎn)而言�����,大多數(shù)治療方法都是面向廣大患者群體使用的�。然而,疾病的病因十分復(fù)雜����,通常由遺傳、環(huán)境和生活方式等相互作用引起�����。因此�����,治療結(jié)果可能差別很大�����,患者可能完全消除或減輕癥狀�����,也有可能面臨著中度或嚴(yán)重的失敗治療�����,產(chǎn)生更多副作用或病情進(jìn)展沒有變化,而精準(zhǔn)醫(yī)療是解決這一問題的潛在方案1,2�����。精準(zhǔn)醫(yī)療的首要目標(biāo)是利用具體病患的分子特征來了解如下多個(gè)方面的信息:
?
● 診斷信息���。該病的特征是什么?
●?患者分層�����。我們能否利用生物標(biāo)志物來完善復(fù)雜疾病的亞型�?
●?疾病預(yù)后。該病有多嚴(yán)重���?它的病程有多快�����?
●?治療選擇�。對(duì)于特定的病患�����,最佳的治療方案是什么?
●?治療應(yīng)答指標(biāo)����。藥物的效果是否符合預(yù)期,還是應(yīng)該改變劑量��,嘗試另一種治療方法����,或者用另一種藥物作為補(bǔ)充?
●?新藥開發(fā)�����。我們能否確定新的治療靶點(diǎn)�?
?
在尋找這些問題答案的過程中,相當(dāng)一部分的研究經(jīng)費(fèi)被用于確定合適的治療靶點(diǎn)和生物標(biāo)志物�����。下面�����,我們將著重介紹當(dāng)前方法����、臨床上的成功案例以及精準(zhǔn)醫(yī)療研究的發(fā)展前景��。
?
精準(zhǔn)醫(yī)療的基本原則
●?基于遺傳變異的方法?
盡管精準(zhǔn)醫(yī)療的概念早在20世紀(jì)60年代就已經(jīng)存在�����,但直到21世紀(jì)初人類基因組計(jì)劃完成后,這一運(yùn)動(dòng)才獲得了真正的推動(dòng)力1�����。這一壯舉使得科學(xué)家們能夠?qū)θ祟愡z傳變異(如突變和單核苷酸多態(tài)性)進(jìn)行大規(guī)模研究��,并確定其與獨(dú)特表型或疾病的關(guān)聯(lián)性3����。這些項(xiàng)目被統(tǒng)稱為全基因組關(guān)聯(lián)分析(GWAS),確定了數(shù)十萬(wàn)種疾病和性狀的風(fēng)險(xiǎn)變異4�����。
在某些情況下�,識(shí)別疾病相關(guān)風(fēng)險(xiǎn)變異揭示了潛在的疾病生物學(xué)機(jī)理,這反過來又能夠推動(dòng)醫(yī)療發(fā)展�。除此之外���,GWAS確定了可選擇的適合患者治療方法的生物標(biāo)志物。下面我們重點(diǎn)介紹幾個(gè)關(guān)鍵應(yīng)用:
案例研究1:克羅恩病和IL-12/IL-23抑制劑
在克羅恩?���。–rohn’s disease)中,GWAS發(fā)現(xiàn)細(xì)胞因子IL-12和IL-23與疾病發(fā)展相關(guān)聯(lián)5��。針對(duì)這些分子促炎作用的基礎(chǔ)研究促進(jìn)了靶向這一途徑的免疫治療藥物實(shí)驗(yàn)的成功6���。?
案例研究2:可用于治療選擇的肺癌生物標(biāo)志物
DNA測(cè)序已經(jīng)發(fā)現(xiàn)了多種肺癌相關(guān)的基因變異7�����,其中7種已被FDA批準(zhǔn)用于治療8���。這使得醫(yī)生可以利用基因檢測(cè)來選擇治療方法,并希望借此改善患者的預(yù)后�。
然而,僅識(shí)別疾病相關(guān)的風(fēng)險(xiǎn)變異可能會(huì)導(dǎo)致不完備的病因?qū)W觀點(diǎn)�����。因此,為了充分利用這些發(fā)現(xiàn)推進(jìn)精準(zhǔn)醫(yī)療的發(fā)展���,必須了解其功能性結(jié)果�。例如�����,基因組中蛋白質(zhì)編碼區(qū)的變異可能影響蛋白質(zhì)的結(jié)構(gòu)��、功能和表達(dá)��。如囊性纖維化疾病中每一種變異都代表著一種不同的分子結(jié)果�����,可能需要以不同的方式對(duì)待�����。?
案例研究3:遺傳風(fēng)險(xiǎn)變異為囊性纖維化的治療提供信息
多種遺傳變異與囊性纖維化有關(guān)�����。根據(jù)致病變異的不同����,兩種對(duì)CFTR蛋白具有不同作用機(jī)制的藥物(幫助運(yùn)輸?shù)郊?xì)胞表面或離子通道功能)可單獨(dú)或聯(lián)合使用9-11。
值得注意的是��,90%以上經(jīng)GWAS確認(rèn)的變異都位于基因組的非編碼區(qū)12�,而非編碼區(qū)富含細(xì)胞類型特異的、能夠激活基因表達(dá)的順式調(diào)控元件(即增強(qiáng)子)13����。因此,許多研究人員專注于研究疾病風(fēng)險(xiǎn)變體如何影響轉(zhuǎn)錄調(diào)節(jié)����,而不是蛋白質(zhì)結(jié)構(gòu)或功能14-16。
目前面臨的挑戰(zhàn)在于了解靶標(biāo)基因是什么�����。非編碼區(qū)可以通過直接和間接的機(jī)制跨越基因組距離發(fā)揮作用���;因此���,與疾病變異最接近的基因不一定是因果決定因素17,18。
?
●?基于基因表達(dá)的方法
研究人員發(fā)現(xiàn)�����,在不同的疾病狀態(tài)下,包括癌癥�����、自身免疫性疾病�����、神經(jīng)退行性疾病����、心血管疾病和其他疾病等,基因表達(dá)都發(fā)生了很大的變化19-23�,這突出了通過全基因組調(diào)查方法發(fā)掘疾病的轉(zhuǎn)錄特征的必要性?����;虮磉_(dá)研究�,包括微陣列和RNA測(cè)序方法在內(nèi)�����,已經(jīng)幫助研究者確定了許多疾病的亞型。
?
案例研究4:乳腺癌患者分層和個(gè)性化治療選擇
歸功于基于精準(zhǔn)醫(yī)療的診斷和治療的應(yīng)用�����,乳腺癌患者的五年生存率已大幅提高24(從1975年的76%提高到現(xiàn)在的90%以上25)���。乳腺癌可以通過多種信號(hào)受體的表達(dá)進(jìn)行分類�����,為診斷����、選擇合適的治療組合和復(fù)發(fā)風(fēng)險(xiǎn)提供信息���。例如��,酪氨酸激酶HER2(HER2+)表達(dá)陽(yáng)性的患者通常使用曲妥珠單抗(trastuzumab)����,一種靶向HER226的單克隆抗體����。將HER2狀態(tài)等分子分類與細(xì)胞分化和增殖的定量測(cè)量相結(jié)合�����,也為患者的預(yù)后提供了信息27����。這些只是大量研究結(jié)果中的其中幾個(gè)例子����,它們推動(dòng)了該領(lǐng)域的發(fā)展,幫助醫(yī)生治療并給予了乳腺癌患者及其家庭支持���。
?
基于基因表達(dá)的個(gè)性化醫(yī)療研究仍在進(jìn)行�����,既要進(jìn)一步完善有既定生物標(biāo)志物的癌癥亞類��,如乳腺癌28�,也要完善沒有生物標(biāo)志物的癌癥亞類�����,如一些腦癌29��。這些發(fā)現(xiàn)將產(chǎn)生新的生物標(biāo)志物和治療靶點(diǎn)��,可能拯救許多生命�。
?
當(dāng)前精準(zhǔn)醫(yī)療方法的缺陷
在上文中我們重點(diǎn)介紹了精準(zhǔn)醫(yī)療領(lǐng)域在多方面的臨床成功案例。盡管精準(zhǔn)醫(yī)療已經(jīng)取得了這些顯著的成就���,但GWAS和轉(zhuǎn)錄組學(xué)往往無(wú)法產(chǎn)生與臨床相關(guān)的生物標(biāo)志物或藥物靶點(diǎn)��,令許多疾病難以診斷和治療����。這可以歸因于許多懸而未決的問題:?
●?哪種變異是疾病的真實(shí)原因�?
這些變異很少單獨(dú)發(fā)生。事實(shí)上����,變異通常是在被稱為單倍型的大塊DNA中成組遺傳的,這使得我們很難知道哪些變異(如果確實(shí)存在的話)可能會(huì)影響疾病狀態(tài)�。
●?遺傳和環(huán)境的影響如何交叉作用?
事實(shí)上�,一些遺傳變異的外顯率受到生活方式30或環(huán)境因素31的調(diào)節(jié),如服用某些處方藥�����、運(yùn)動(dòng)和環(huán)境污染,其基因調(diào)控影響和疾病風(fēng)險(xiǎn)與暴露程度相關(guān)����。
●?環(huán)境因素是否更可能是主要原因?
一些與疾病相關(guān)的基因調(diào)控變化與遺傳變異完全無(wú)關(guān)��。相反�,這些基因調(diào)節(jié)變化的根本原因——與從過敏32到低出生體重33到心臟病34等狀態(tài)相關(guān)——取決于環(huán)境暴露。
●?基因表達(dá)是否涵蓋了我們需要知道的一切�����?
目前研究異常RNA表達(dá)的方法提供了細(xì)胞內(nèi)當(dāng)前情況的快照����。它們不能提供有關(guān)細(xì)胞“準(zhǔn)備做什么”的信息,如治療引起的遺傳程序的變化或抗藥性的發(fā)展�。這種機(jī)制信息的缺乏留下了許多關(guān)于治療應(yīng)答和復(fù)發(fā)可能性的開放性問題,而這些問題對(duì)個(gè)性化護(hù)理具有極大的參考價(jià)值����。
●?為什么我們只關(guān)注蛋白質(zhì)編碼區(qū)?
基因表達(dá)和遺傳變異是疾病的主要指標(biāo)——但它們只是影響因素的一部分��。事實(shí)上,人類基因組中只有不到1%編碼蛋白質(zhì)35�?��;蛘{(diào)控主要由非編碼區(qū)控制���,非編碼區(qū)可以作為增強(qiáng)子,形成染色質(zhì)環(huán)��,結(jié)合轉(zhuǎn)錄因子�,或招募表觀遺傳修飾酶和閱讀器蛋白。此外���,許多疾病風(fēng)險(xiǎn)變異也富集在非編碼DNA中����。因此�����,非編碼DNA和基于染色質(zhì)的機(jī)制代表了大量未開發(fā)的生物標(biāo)志物和藥物靶點(diǎn)�����。
?
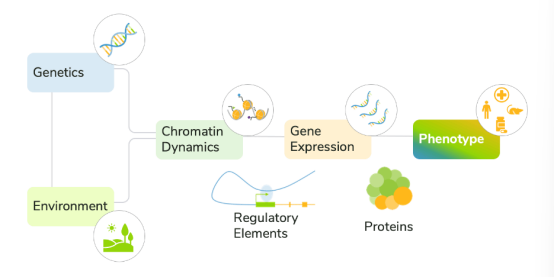
遺傳�����、環(huán)境、轉(zhuǎn)錄和染色質(zhì)的融合使得疾病表型發(fā)展十分復(fù)雜(Figure?1)���。目前的方法通常無(wú)法考慮這些不同的因素����,使得人們難以預(yù)測(cè)疾病進(jìn)展并對(duì)治療作出及時(shí)的反應(yīng)�。
 ?
?
Figure 1?Environmental and molecular factors together drive disease development.
?
將表觀基因組學(xué)納入精準(zhǔn)醫(yī)療
表觀基因組可能是確定新型生物標(biāo)志物和藥物靶點(diǎn)以推進(jìn)精準(zhǔn)醫(yī)療的關(guān)鍵。表觀基因組包括復(fù)雜的分子代碼���,能夠調(diào)節(jié)染色質(zhì)結(jié)構(gòu)和功能以控制基因表達(dá)�。因此��,對(duì)表觀基因組進(jìn)行的研究可以作為一個(gè)重要的分子工具�,幫助我們了解由遺傳變異、環(huán)境和治療導(dǎo)致的基因表達(dá)變化����。
為此,染色質(zhì)修飾�,包括DNA甲基化和組蛋白翻譯后修飾(PTMs),已經(jīng)為基因調(diào)控程序提供了顯著的見解,并正在成為精準(zhǔn)醫(yī)療研究的重要組成部分���。
?
●?DNA甲基化
從歷史觀點(diǎn)上說��,繪制DNA甲基化圖譜已經(jīng)成為表觀基因組的精準(zhǔn)醫(yī)療研究的首選方式。這種既定的抑制性染色質(zhì)標(biāo)記有多種檢測(cè)選擇:甲基化敏感的PCR和限制性酶���、基于抗體的方法(如MeDIP)或亞硫酸氫鹽轉(zhuǎn)化后的測(cè)序(BS-seq/WGBS)36,37��。DNA甲基化與診斷學(xué)的結(jié)合帶來了令人興奮的進(jìn)展:
?
案例研究5:中樞神經(jīng)系統(tǒng)(CNS)腫瘤的分類和治療選擇
許多原發(fā)性中樞神經(jīng)系統(tǒng)腫瘤難以用傳統(tǒng)的組織學(xué)方法進(jìn)行分類38��。例如�,中樞神經(jīng)系統(tǒng)神經(jīng)外胚層腫瘤是類別廣泛�����,其組織學(xué)特征是小的����、分化不良的神經(jīng)元和神經(jīng)膠質(zhì)。在一項(xiàng)研究中���,DNA甲基化分析的整合將75%以上的腫瘤重新分類為更具體的亞型�,如髓母細(xì)胞瘤、高級(jí)膠質(zhì)瘤���、室管膜瘤和松果體瘤39����。在其他原發(fā)性中樞神經(jīng)系統(tǒng)腫瘤中��,如膠質(zhì)母細(xì)胞瘤�,選擇性啟動(dòng)子(如MGMT)的甲基化促進(jìn)治療的靈敏度,如替莫唑胺(temozolomide)�,指導(dǎo)治療選擇40。
?
案例研究6:無(wú)創(chuàng)體外診斷的發(fā)展
在早期研究中���,在患者血漿或血清中的循環(huán)無(wú)細(xì)胞核小體上能夠檢測(cè)到DNA甲基化�,并在一些概念驗(yàn)證的臨床研究中觀察到���,如肺癌���、乳腺癌和結(jié)直腸癌37,41-43。這為開發(fā)FDA批準(zhǔn)的體外診斷測(cè)試鋪平了道路���,這些測(cè)試來自容易獲得的樣本���,如Cologuard?和Epi proColon?的糞便或血液���,以及幾種有前景的液體活檢測(cè)試,包括GRAIL的Galleri?����,以檢測(cè)癌癥,無(wú)論其類型如何44-46��。
?
然而�,由于無(wú)法提供不同染色質(zhì)區(qū)間的細(xì)節(jié)���,特別是組織/細(xì)胞特異性增強(qiáng)子的活性��,DNA甲基化的研究對(duì)某些應(yīng)用來說是不夠的�。此外��,DNA甲基化并不總是一種抑制性標(biāo)記47�����,這意味著它對(duì)基因表達(dá)的影響可能是模糊的�。雖然DNA甲基化已經(jīng)證明破譯表觀基因組可以為精準(zhǔn)醫(yī)療方法提供信息�����,但就揭示表觀基因組學(xué)和染色質(zhì)上的基因表達(dá)修飾機(jī)制而言��,這只是冰山一角�����。
?
組蛋白翻譯后修飾?
染色質(zhì)上顯著的化學(xué)和蛋白質(zhì)多樣性充滿了潛在的精準(zhǔn)醫(yī)療靶點(diǎn)���。具體而言,組蛋白PTMs和染色質(zhì)結(jié)合蛋白(如轉(zhuǎn)錄因子)對(duì)基因表達(dá)產(chǎn)生了強(qiáng)大而動(dòng)態(tài)的影響�����。迄今為止��,超過100種獨(dú)特的組蛋白PTMs或其組合被認(rèn)為與人類疾病有關(guān)����,包括多種癌癥48-53,與此同時(shí)人們也發(fā)現(xiàn)組蛋白PTM模式的整體改變可以預(yù)測(cè)疾病狀態(tài)�����、復(fù)發(fā)情況和患者的治療應(yīng)答54-56。
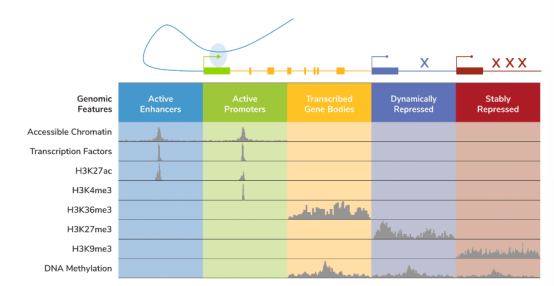
與DNA甲基化甚至染色質(zhì)可及性檢測(cè)(如ATAC-seq)不同�����,組蛋白PTMs提供了對(duì)不同���、特定基因組特征的認(rèn)知��。全基因組染色質(zhì)圖譜工具的開發(fā)有助于將組蛋白PTMs分配到特定的基因組區(qū)間(Figure?2)����,如活躍的增強(qiáng)子(H3K27ac)����、活躍的啟動(dòng)子(H3K4me3)�����、活躍的基因體(H3K36me3)���,甚至被抑制的基因(H3K9me2/3���,H3K27me3)�����。因此����,研究組蛋白PTMs可以闡明基因調(diào)控格局的多樣性���,包括細(xì)胞的功能����。一個(gè)典型的例子是二價(jià)H3K4me3/H3K27me3標(biāo)記���,它表示未分化的干細(xì)胞或原生細(xì)胞中 '準(zhǔn)備好'激活的增強(qiáng)子和啟動(dòng)子57��。值得注意的是���,在癌癥中經(jīng)常觀察到二價(jià)基因表達(dá)的改變58,它們可能與病情惡化59或耐藥性60有關(guān)�����。
 ?
?
Figure 2?Overview of the localization of chromatin features.?
因此��,基因組圖譜研究包含了個(gè)性化醫(yī)療的關(guān)鍵信息。這一領(lǐng)域的前沿研究已經(jīng)在精準(zhǔn)醫(yī)療領(lǐng)域產(chǎn)生了影響:
?
案例研究7:癌癥治療反應(yīng)的預(yù)測(cè)和調(diào)節(jié)
一項(xiàng)研究發(fā)現(xiàn)�,組蛋白PTM H3K27ac是增強(qiáng)子和活躍轉(zhuǎn)錄程序的標(biāo)志物,可用于區(qū)分結(jié)直腸癌的亞類型�����,包括預(yù)后結(jié)果和治療應(yīng)答61��。此外�,使用這些增強(qiáng)子亞型分類的新型組合療法在患者衍生的異種移植中顯示出療效。另一項(xiàng)研究發(fā)現(xiàn)���,抑制性組蛋白PTM H3K27me3使乳腺癌細(xì)胞對(duì)化療敏感�����,而阻止這一標(biāo)記會(huì)抑制藥物耐受性并延緩腫瘤形成60,這表明組蛋白PTMs具有治療潛力����。
?
這些研究共同表明,表觀基因組研究具有指導(dǎo)臨床決策和藥物開發(fā)的強(qiáng)大潛力�����。
??
與整合表觀基因組學(xué)有關(guān)的挑戰(zhàn)
雖然染色質(zhì)圖譜展示了精準(zhǔn)醫(yī)療應(yīng)用的巨大前景,但表觀基因組在臨床應(yīng)用方面的研究一直極具挑戰(zhàn)性����。
?
歷史上,染色質(zhì)圖譜是通過ChIP-seq進(jìn)行的�。雖然具有突破性,但也有很大的局限性��,這種局限阻礙了它在精準(zhǔn)醫(yī)療中的應(yīng)用��。具體來說���,這些限制包括:
?
●?低靈敏度和低通量�,需要數(shù)以百萬(wàn)計(jì)的細(xì)胞�,深度測(cè)序,以及大量的實(shí)踐時(shí)間�。
●?難以適應(yīng)不同的細(xì)胞類型,需要對(duì)每個(gè)新的細(xì)胞類型進(jìn)行優(yōu)化��。
●?組蛋白PTMs的非特異性抗體——事實(shí)上����,絕大多數(shù)(如EpiCypher62所示,>70%)與相關(guān)標(biāo)記發(fā)生交叉反應(yīng)。
●?所有這些都將導(dǎo)致高成本和低質(zhì)量的數(shù)據(jù)��!
?
這些挑戰(zhàn)共同限制了ChIP-seq數(shù)據(jù)的規(guī)模���、應(yīng)用和解讀�。ChIP的可靠性差���,也使得它難以研究疾病的異質(zhì)性和患者的特定治療應(yīng)答�����。
總之��,ChIP-seq無(wú)法完成表觀基因組學(xué)在精準(zhǔn)醫(yī)療方面所需的工作��。那么還有什么其他選擇嗎���?
?
CUT&RUN和CUT&Tag:改進(jìn)的精準(zhǔn)醫(yī)療表觀基因組學(xué)研究工具
CUT&RUN與CUT&Tag分析解決了許多與ChIP-seq相關(guān)的問題,只需取用少量細(xì)胞和測(cè)序讀數(shù)就能生成高分辨率的圖譜�,并且十分快速——EpiCypher的CUTANA? CUT&RUN與CUT&Tag分析法從細(xì)胞到測(cè)序文庫(kù)只需2到3天,而ChIP需要一周(或更長(zhǎng)時(shí)間)�����。
?
CUT&RUN和CUT&Tag是研究的理想選擇�,可以提高我們對(duì)疾病風(fēng)險(xiǎn)變異、基因調(diào)控程序和診斷/治療生物標(biāo)志物的認(rèn)識(shí)����。與ChIP-seq相比,CUT&RUN和CUT&Tag與個(gè)性化醫(yī)學(xué)應(yīng)用相關(guān)的優(yōu)勢(shì)和特點(diǎn)包括:
?
●?高靈敏度和高通量:經(jīng)過簡(jiǎn)化的工作流程��,檢測(cè)只需要5,000個(gè)細(xì)胞
●?廣泛的細(xì)胞類型兼容性:干細(xì)胞�����、免疫細(xì)胞�����、患者衍生的異種細(xì)胞����、FACS分選的細(xì)胞以及小鼠和人類原代細(xì)胞、冷凍樣品和輕度交聯(lián)的材料
●?經(jīng)驗(yàn)證的抗體����,以確保您對(duì)結(jié)果的信賴
●?成本低——與ChIP-seq相比,每個(gè)反應(yīng)大約減少70%的成本��。
?
CUT&RUN對(duì)大多數(shù)靶標(biāo)都很穩(wěn)健,包括瞬時(shí)相互作用蛋白(如染色質(zhì)重塑酶)��、先驅(qū)因子����、異染色質(zhì)靶點(diǎn),并具有單細(xì)胞兼容試驗(yàn)方案63��,能分析異質(zhì)組織��。重要的是���,CUT&Tag只推薦用于組蛋白PTM靶點(diǎn)���。
許多課題組已經(jīng)使用EpiCypher的CUT&RUN64-67和CUT&Tag68,69檢測(cè)來研究染色質(zhì)結(jié)構(gòu)并用于生物醫(yī)學(xué)研究。其他文獻(xiàn)引用已經(jīng)證明了這項(xiàng)技術(shù)在精準(zhǔn)醫(yī)療方面的前景:
?
案例研究8:神經(jīng)元中與疾病相關(guān)的增強(qiáng)子鑒定
通過結(jié)合H3K27ac CUT&RUN����、ATAC-seq和RNA-seq,一組便能夠識(shí)別興奮性與抑制性人類神經(jīng)元特有的增強(qiáng)子70��。增強(qiáng)子圖譜與精神疾病風(fēng)險(xiǎn)變異數(shù)據(jù)庫(kù)相結(jié)合���,顯示出與精神分裂癥�����、多動(dòng)癥和雙相情感障礙有關(guān)的變異富集���。這項(xiàng)研究直接證明了表觀基因組學(xué)與現(xiàn)有數(shù)據(jù)集的整合可以進(jìn)一步了解疾病風(fēng)險(xiǎn)變異體的功能和異常基因的表達(dá)�����,從而推動(dòng)精準(zhǔn)醫(yī)療���。
?
案例研究9:液體活檢轉(zhuǎn)錄印跡的鑒定
CUT&RUN已被用于識(shí)別異種移植中ER+乳腺癌的轉(zhuǎn)錄印跡71��。轉(zhuǎn)錄圖譜被用來對(duì)人類乳腺腫瘤進(jìn)行亞分類�����,而且值得注意的是���,在患者的血漿中也能檢測(cè)到71。這些研究直接證明了二代CUT&RUN基因組圖譜技術(shù)在推進(jìn)新興的無(wú)創(chuàng)液體活檢個(gè)性化醫(yī)學(xué)應(yīng)用方面的潛力�。
?
結(jié)論和展望
盡管精準(zhǔn)醫(yī)療面臨著定義患者特異性分子特征的困難,不可否認(rèn)它是一個(gè)令人振奮的領(lǐng)域�����。用于精準(zhǔn)醫(yī)療的染色質(zhì)研究有可能在兩個(gè)主要方面顯著改變?cè)擃I(lǐng)域:
?
1.?新型染色質(zhì)生物標(biāo)志物的鑒定和驗(yàn)證——在現(xiàn)有的基因組和轉(zhuǎn)錄組方法的基礎(chǔ)上展現(xiàn)了一個(gè)需要監(jiān)管的、環(huán)境敏感的尚未開發(fā)領(lǐng)域���。
2.?闡明驅(qū)動(dòng)疾病的轉(zhuǎn)錄調(diào)控機(jī)制——這將有助于表征疾病風(fēng)險(xiǎn)變異��,并確定新的轉(zhuǎn)錄生物標(biāo)志物和藥物靶點(diǎn)��。
?
CUT&RUN與CUT&Tag是能夠用于實(shí)現(xiàn)這些目標(biāo)的強(qiáng)大檢測(cè)方法�����。表觀基因組學(xué)在精準(zhǔn)醫(yī)療中的其他應(yīng)用包括精細(xì)繪制獨(dú)特細(xì)胞類型中的染色質(zhì)區(qū)室�����,以幫助闡明疾病發(fā)展和進(jìn)展的染色質(zhì)調(diào)節(jié)機(jī)制��。這種表觀基因組圖譜可與其他測(cè)序方式相結(jié)合����,包括DNA甲基化���、RNA-seq��、ATAC-seq或Hi-C�����,以表征個(gè)別患者的疾病特征并改善臨床預(yù)后�����。
?
?
參考文獻(xiàn)
1.?J?rgensen JT. Twenty Years with Personalized Medicine: Past, Present, and Future of Individualized Pharmacotherapy. Oncologist 24, e432-e40 (2019). PubMed PMID: 30940745.
2.?Ashley EA. Towards precision medicine. Nat Rev Genet 17, 507-22 (2016). PubMed PMID: 27528417.
3.?Ikegawa S. A short history of the genome-wide association study: where we were and where we are going. Genomics Inform 10, 220-5 (2012). PubMed PMID: 23346033.
4.?Uffelmann E et al. Genome-wide association studies. Nature Reviews Methods Primers 1, 59 (2021).?https://doi.org/10.1038/s43586-021-00056-9.
5.?Wang K et al. Diverse genome-wide association studies associate the IL12/IL23 pathway with Crohn Disease. Am J Hum Genet 84, 399-405 (2009). PubMed PMID: 19249008.
6.?Moschen AR et al. IL-12, IL-23 and IL-17 in IBD: immunobiology and therapeutic targeting. Nat Rev Gastroenterol Hepatol 16, 185-96 (2019). PubMed PMID: 30478416.
7.?Greulich H. The genomics of lung adenocarcinoma: opportunities for targeted therapies. Genes Cancer 1, 1200-10 (2010). PubMed PMID: 21779443.
8.?Targeted Therapies for Lung Cancer: American Lung Association. Available from:?https://www.lung.org/lung-health-diseases/lung-disease-lookup/lung-cancer/treatment/types-of-treatment/targeted-therapies.
9.?Boyle MP et al. A CFTR corrector (lumacaftor) and a CFTR potentiator (ivacaftor) for treatment of patients with cystic fibrosis who have a phe508del CFTR mutation: a phase 2 randomised controlled trial. Lancet Respir Med 2, 527-38 (2014). PubMed PMID: 24973281.
10.?Condren ME et al. Ivacaftor: a novel gene-based therapeutic approach for cystic fibrosis. J Pediatr Pharmacol Ther 18, 8-13 (2013). PubMed PMID: 23616732.
11.?Wainwright CE et al. Lumacaftor-Ivacaftor in Patients with Cystic Fibrosis Homozygous for Phe508del CFTR. N Engl J Med 373, 1783-4 (2015). PubMed PMID: 26510034.
12.?Edwards SL et al. Beyond GWASs: illuminating the dark road from association to function. Am J Hum Genet 93, 779-97 (2013). PubMed PMID: 24210251.
13.?Gasperini M et al. Towards a comprehensive catalogue of validated and target-linked human enhancers. Nat Rev Genet 21, 292-310 (2020). PubMed PMID: 31988385.
14.?Morley M et al. Genetic analysis of genome-wide variation in human gene expression. Nature 430, 743-7 (2004). PubMed PMID: 15269782.
15.?Schadt EE et al. An integrative genomics approach to infer causal associations between gene expression and disease. Nat Genet 37, 710-7 (2005). PubMed PMID: 15965475.
16.?Emilsson V et al. Genetics of gene expression and its effect on disease. Nature 452, 423-8 (2008). PubMed PMID: 18344981.
17.?McGovern A et al. Capture Hi-C identifies a novel causal gene, IL20RA, in the pan-autoimmune genetic susceptibility region 6q23. Genome Biol 17, 212 (2016). PubMed PMID: 27799070.
18.?Smemo S et al. Obesity-associated variants within FTO form long-range functional connections with IRX3. Nature 507, 371-5 (2014). PubMed PMID: 24646999.
19.?Bradner JE et al. Transcriptional Addiction in Cancer. Cell 168, 629-43 (2017). PubMed PMID: 28187285.
20.?Nagafuchi Y et al. Lessons From Transcriptome Analysis of Autoimmune Diseases. Front Immunol 13, 857269 (2022). PubMed PMID: 35663941.
21.?Cooper-Knock J et al. Gene expression profiling in human neurodegenerative disease. Nat Rev Neurol 8, 518-30 (2012). PubMed PMID: 22890216.
22.?Liu Y et al. RNA-Seq identifies novel myocardial gene expression signatures of heart failure. Genomics 105, 83-9 (2015). PubMed PMID: 25528681.
23.?Lee TI et al. Transcriptional regulation and its misregulation in disease. Cell 152, 1237-51 (2013). PubMed PMID: 23498934.
24.?Siegel RL et al. Cancer statistics, 2022. CA Cancer J Clin 72, 7-33 (2022). PubMed PMID: 35020204.
25.?Cancer Stat Facts: Female Breast Cancer: National Cancer Institute. Available from:?https://seer.cancer.gov/statfacts/html/breast.html.
26.?Slamon DJ et al. Use of chemotherapy plus a monoclonal antibody against HER2 for metastatic breast cancer that overexpresses HER2. N Engl J Med 344, 783-92 (2001). PubMed PMID: 11248153.
27.?Sotiriou C et al. Gene-expression signatures in breast cancer. N Engl J Med 360, 790-800 (2009). PubMed PMID: 19228622.
28.?Zhao W et al. SCD5 expression correlates with prognosis and response to neoadjuvant chemotherapy in breast cancer. Sci Rep 11, 8976 (2021). PubMed PMID: 33903614.
29.?Yuan Q et al. Identifying Differential Expression Genes and Prognostic Signature Based on Subventricular Zone Involved Glioblastoma. Front Genet 13, 912227 (2022). PubMed PMID: 35873494.
30.?Knowles DA et al. Allele-specific expression reveals interactions between genetic variation and environment. Nat Methods 14, 699-702 (2017). PubMed PMID: 28530654.
31.?Favé MJ et al. Gene-by-environment interactions in urban populations modulate risk phenotypes. Nat Commun 9, 827 (2018). PubMed PMID: 29511166.
32.?Prescott S et al. The role of epigenetic dysregulation in the epidemic of allergic disease. Clin Epigenetics 2, 223-32 (2011). PubMed PMID: 21949548.
33.?Heijmans BT et al. Persistent epigenetic differences associated with prenatal exposure to famine in humans. Proc Natl Acad Sci U S A 105, 17046-9 (2008). PubMed PMID: 18955703.
34.?Breitling LP et al. Smoking, F2RL3 methylation, and prognosis in stable coronary heart disease. Eur Heart J 33, 2841-8 (2012). PubMed PMID: 22511653.
35.?Sheffield NC et al. Identifying and characterizing regulatory sequences in the human genome with chromatin accessibility assays. Genes (Basel) 3, 651-70 (2012). PubMed PMID: 24705081.
36.?Mattei AL et al. DNA methylation: a historical perspective. Trends Genet 38, 676-707 (2022). PubMed PMID: 35504755.
37.?Laird PW. The power and the promise of DNA methylation markers. Nat Rev Cancer 3, 253-66 (2003). PubMed PMID: 12671664.
38.?Galbraith K et al. DNA methylation as a diagnostic tool. Acta Neuropathol Commun 10, 71 (2022). PubMed PMID: 35527288.
39.?Sturm D et al. New Brain Tumor Entities Emerge from Molecular Classification of CNS-PNETs. Cell 164, 1060-72 (2016). PubMed PMID: 26919435.
40.?Butler M et al. MGMT Status as a Clinical Biomarker in Glioblastoma. Trends Cancer 6, 380-91 (2020). PubMed PMID: 32348734.
41.?Li P et al. Liquid biopsies based on DNA methylation as biomarkers for the detection and prognosis of lung cancer. Clin Epigenetics 14, 118 (2022). PubMed PMID: 36153611.
42.?Manoochehri M et al. DNA methylation biomarkers for noninvasive detection of triple-negative breast cancer using liquid biopsy. Int J Cancer 152, 1025-35 (2023). PubMed PMID: 36305646.
43.?Jin S et al. Efficient detection and post-surgical monitoring of colon cancer with a multi-marker DNA methylation liquid biopsy. Proc Natl Acad Sci U S A 118, (2021). PubMed PMID: 33495330.
44.?Pickhardt PJ. Emerging stool-based and blood-based non-invasive DNA tests for colorectal cancer screening: the importance of cancer prevention in addition to cancer detection. Abdom Radiol (NY) 41, 1441-4 (2016). PubMed PMID: 27259335.
45.?Nadauld LD et al. The PATHFINDER Study: Assessment of the Implementation of an Investigational Multi-Cancer Early Detection Test into Clinical Practice. Cancers (Basel) 13, (2021). PubMed PMID: 34298717.
46.?Taryma-Le?niak O et al. Current status of development of methylation biomarkers for in vitro diagnostic IVD applications. Clin Epigenetics 12, 100 (2020). PubMed PMID: 32631437.
47.?Rauluseviciute I et al. DNA hypermethylation associated with upregulated gene expression in prostate cancer demonstrates the diversity of epigenetic regulation. BMC Med Genomics 13, 6 (2020). PubMed PMID: 31914996.
48.?Chopra M et al. Disturbing the histone code in leukemia: translocations and mutations affecting histone methyl transferases. Cancer Genet 208, 192-205 (2015). PubMed PMID: 25592767.
49.?Greenblatt SM et al. Chromatin modifiers and the promise of epigenetic therapy in acute leukemia. Leukemia 28, 1396-406 (2014). PubMed PMID: 24609046.
50.?Gajer JM et al. Histone acetyltransferase inhibitors block neuroblastoma cell growth in vivo. Oncogenesis 4, e137 (2015). PubMed PMID: 25664930.
51.?Witt O et al. Targeting histone deacetylases in neuroblastoma. Curr Pharm Des 15, 436-47 (2009). PubMed PMID: 19199971.
52.?Hanmod SS et al. Targeting histone deacetylases (HDACs) and Wee1 for treating high-risk neuroblastoma. Pediatr Blood Cancer 62, 52-9 (2015). PubMed PMID: 25308916.
53.?Kobayashi K et al. Epigenetic regulation of the neuroblastoma genes, Arid3b and Mycn. Oncogene 32, 2640-8 (2013). PubMed PMID: 22751132.
54.?Seligson DB et al. Global histone modification patterns predict risk of prostate cancer recurrence. Nature 435, 1262-6 (2005). PubMed PMID: 15988529.
55.?Kamińska K et al. Prognostic and Predictive Epigenetic Biomarkers in Oncology. Mol Diagn Ther 23, 83-95 (2019). PubMed PMID: 30523565.
56.?Seligson DB et al. Global levels of histone modifications predict prognosis in different cancers. Am J Pathol 174, 1619-28 (2009). PubMed PMID: 19349354.
57.?Macrae TA et al. Regulation, functions and transmission of bivalent chromatin during mammalian development. Nat Rev Mol Cell Biol 24, 6-26 (2023). PubMed PMID: 36028557.
58.?Kumar D et al. Decoding the function of bivalent chromatin in development and cancer. Genome Res 31, 2170-84 (2021). PubMed PMID: 34667120.
59.?Chaffer CL et al. Poised chromatin at the ZEB1 promoter enables breast cancer cell plasticity and enhances tumorigenicity. Cell 154, 61-74 (2013). PubMed PMID: 23827675.
60.?Marsolier J et al. H3K27me3 conditions chemotolerance in triple-negative breast cancer. Nat Genet 54, 459-68 (2022). PubMed PMID: 35410383.
61.?Orouji E et al. Chromatin state dynamics confers specific therapeutic strategies in enhancer subtypes of colorectal cancer. Gut 71, 938-49 (2022). PubMed PMID: 34059508.
62.?Shah RN et al. Examining the Roles of H3K4 Methylation States with Systematically Characterized Antibodies. Mol Cell 72, 162-77.e7 (2018). PubMed PMID: 30244833.
63.?Hainer SJ et al. Profiling of Pluripotency Factors in Single Cells and Early Embryos. Cell 177, 1319-29.e11 (2019). PubMed PMID: 30955888.
64.?Janssens DH et al. Automated in situ chromatin profiling efficiently resolves cell types and gene regulatory programs. Epigenetics Chromatin 11, 74 (2018). PubMed PMID: 30577869.
65.?Theisen ER et al. Chromatin profiling reveals relocalization of lysine-specific demethylase 1 by an oncogenic fusion protein. Epigenetics 16, 405-24 (2021). PubMed PMID: 32842875.
66.?Garcia-Martinez L et al. Endocrine resistance and breast cancer plasticity are controlled by CoREST. Nat Struct Mol Biol 29, 1122-35 (2022). PubMed PMID: 36344844.
67.?Dai X et al. Massively parallel knock-in engineering of human T cells. Nat Biotechnol (2023). PubMed PMID: 36702900.
68.?Sparbier CE et al. Targeting Menin disrupts the KMT2A/B and polycomb balance to paradoxically activate bivalent genes. Nat Cell Biol 25, 258-72 (2023). PubMed PMID: 36635503.
69.?Battistello E et al. Stepwise activities of mSWI/SNF family chromatin remodeling complexes direct T cell activation and exhaustion. Mol Cell 83, 1216-36.e12 (2023). PubMed PMID: 36944333.
70.?Sanchez-Priego C et al. Mapping cis-regulatory elements in human neurons links psychiatric disease heritability and activity-regulated transcriptional programs. Cell Rep 39, 110877 (2022). PubMed PMID: 35649373.
71.?Rao S et al. Transcription factor-nucleosome dynamics from plasma cfDNA identifies ER-driven states in breast cancer. Sci Adv 8, eabm4358 (2022). PubMed PMID: 36001652.
?

更多產(chǎn)品詳情����,請(qǐng)聯(lián)系EpiCypher全國(guó)授權(quán)代理-欣博盛生物?
全國(guó)服務(wù)熱線: 4006-800-892? ? ? ?郵箱: market@neobioscience.com?
深圳: 0755-26755892? ? ? ? ?北京: 010-88594029? ? ? ? ? ??
廣州:020-87615159? ? ? ? ? ?上海: 021-34613729
代理品牌網(wǎng)站: www.yuebanme.com?
自主品牌網(wǎng)站: www.neobioscience.net